Metabolomika jako potencjalna metoda diagnostyczna w medycynie
Autor: Adam Ząbek, Piotr Młynarz*
Wprowadzenie
Utrzymywanie wewnętrznej równowagi układu biologicznego jest jednym z kluczowych zadań organizmu związanych z jego reakcją na bodźce zewnętrzne bądź wewnętrzne. Równowaga zachowań układu biologicznego, mieszcząca się w pewnych zakresach tolerancji określana jest mianem homeostazy dynamicznej, a więc zmieniającej się w czasie [1].
Zrozumienie istoty homeostazy może być bardzo ważne dla głębszego poznania procesów samoorganizacji komórki i całego układu biologicznego jakim jest człowiek. Bardzo często wytrącenie organizmu ze stanu równowagi może wiązać się z zapoczątkowaniem procesu chorobotwórczego. Z tego względu umiejętność mierzenia tych zmian może istotnie przyczynić się do wcześniejszego wykrywania choroby, zanim będą widoczne i odczuwalne jej objawy [2]. Niestety w wielu przypadkach interwencja lekarska pojawia się zbyt późno, kiedy choroba jest już zaawansowana, przez co w konsekwencji leczenie pacjenta jest utrudnione i wielokrotnie mało skuteczne (rys. 1.).
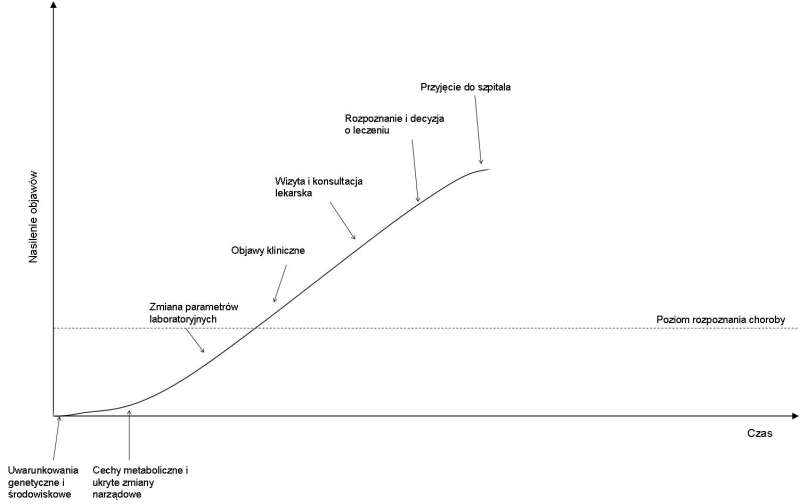
Rys. 1. Wykres zależności nasilenia objawów choroby od szybkości diagnozy [3]
Jeszcze nie tak dawno sądzono, że pomocne w prowadzeniu skutecznej diagnostyki, leczeniu i poznaniu przyczyn różnych chorób będą badania nad rozszyfrowaniem genomu człowieka (genomika) [4]. Wraz z postępem wiedzy okazało się jednak, że sama informacja genetyczna oraz badania komponentu białkowego – proteomu (proteomika), powstającego na jej bazie nie wystarczają do identyfikacji zmian zachodzących w komórce. Istnieje bowiem jeszcze jedna, bardzo istotna sfera komórki, obejmująca zbiór drobnocząsteczkowych związków – metabolitów o różnych właściwościach fizykochemicznych i biochemicznych, pełniących ważne funkcje w organizmie. Na tej podstawie rozwinęła się kolejna z technik „omicznych”, metabolomika, będąca uzupełnieniem badań nad genomem i proteomem [5]. Dopiero takie całościowe, skorelowane ze sobą, spojrzenie na układ biologiczny jakim jest człowiek może prowadzić do poznania mechanizmów jego działania i wyjaśnienia przyczyn wielu chorób.
Metabolomika
Metabolomika to dziedzina nauki, zaliczana do biologii systemowej, obejmująca bardzo szeroki, interdyscyplinarny zakres wiedzy, skupiający się na próbie wyjaśnienia zmian zachodzących w profilu metabolicznym. Na profil ten składają się różnorodne, niskocząsteczkowe związki (w zależności od źródła >1000 lub 1500 Da) takie jak lipidy, kwasy organiczne, węglowodany, aminokwasy, nukleotydy czy też sterydy. W ogólnodostępnej bazie danych metabolitów człowieka HMDB (Human Metabolomics Database) zebrano informacje o ok. 8000 takich związkach [6].
Idea badania metabolitów nie jest nowa, często bowiem wykorzystuje się ją w diagnostyce medycznej. Dotychczasowe jednak podejście wiąże się z rozpoznawaniem choroby na podstawie jednego bądź kilku metabolitów będących charakterystycznymi markerami. Okazuje się natomiast, że w momencie wybicia układu (komórki, tkanki, narządu, organizmu) z homeostazy dynamicznej widoczne są zmiany ilościowe i jakościowe w ogólnym zbiorze metabolitów płynów ustrojowych. Zadaniem metabolomiki jest więc między innymi poszukiwanie tych dyskretnych zmian w całościowym profilu metabolicznym, która w konsekwencji może prowadzić do określania biomarkerów chorobowych, pojawiających się zaraz na początku stanu chorobowego. Takie postępowanie może znacznie pomóc w diagnozie, wyborze efektywnej terapii oraz monitorowaniu przebiegu choroby i leczenia [7].
W badaniach metabolomicznych wykorzystuje się głównie płyny ustrojowe człowieka lub ekstrakty uzyskane z komórek bądź tkanek [7]. Płyny ustrojowe takie jak surowica krwi, osocze, ślina, mocz są szczególnie łatwo dostępne, co sprawia, że metabolomika jako metoda diagnostyczna jest mało inwazyjna. Jednakże badania na ekstraktach komórkowo-tkankowych czy też innych trudniej dostępnych płynach jak np. płyn mózgowo-rdzeniowy jest również bardzo istotne w poszukiwaniu specyficznych biomarkerów chorobowych [7].
Analizę profilu metabolicznego przeprowadza się najczęściej za pomocą spektroskopii magnetycznego rezonansu jądrowego (nuclear magnetic resonance, NMR) oraz spektrometrii mas (mass spectrometry, MS).
W ogólnym ujęciu spektroskopia magnetycznego rezonansu jądrowego NMR umożliwia, za pomocą fal elektromagnetycznych, detekcję jąder atomów 1H, 13C, 15N, 31P i innych, w molekułach znajdujących się w badanym roztworze np. płynach ustrojowych [8]. Każdy z wygenerowanych sygnałów, posiada takie parametry jak przesunięcie chemiczne (ppm, parts per milion), multipletowość, stałą sprzężenia, integrację, które zawierają informacje o strukturze, konformacji oraz dynamice poszczególnych związków chemicznych. Identyfikacja poszczególnych metabolitów może odbywać się w dwojaki sposób. Pierwszy z nich polega na wykorzystaniu widm jednowymiarowych 1D NMR i porównywaniu wartości przesunięcia chemicznego oraz innych parametrów widma poszukiwanego metabolitu z danymi (widmami) z bazy danych, zawierającą widma niskocząsteczkowych związków [8]. Drugi sposób analizy jakościowej polega na określaniu struktur poszczególnych metabolitów na podstawie analizy widm dwuwymiarowych 2D [8].
Bardzo czułą (pozwala oznaczać stężenia związków chemicznych na poziomie nanomolowym), a jednocześnie wydajną metodą stosowaną w jakościowym i ilościowym oznaczaniu metabolitów jest spektrometria mas, MS [9]. Bardzo duży wpływ na zdolność analizy niskocząsteczkowych związków w złożonych próbach biologicznych ma wspomaganie spektrometrii mas preparatywnymi technikami rozdziału, takimi jak chromatografia gazowa (gas chromatography, GC) i cieczowa (liquid chromatography, LC, High Performance Liquid Chromatography,HPLC i Ultra Performance Liquid Chromatography, UPLC) [10]. Technika GC-MS charakteryzuje się bardzo dobrą zdolnością rozdzielczą i powtarzalnością, jednakże wymaga dodatkowego etapu derywatyzacji cząsteczek, w celu zwiększania ich lotności [10]. W przeciwieństwie do metody GC-MS w chromatografii cieczowej LC-MS analizowane próbki rzadko poddaje się derywatyzacji [11-12].
Interpretacja widm masowych, zawierających sygnały przypisane do wartości stosunku mas do ładunku (m/z) zjonizowanych cząsteczek oraz ich fragmentów opiera się na analizie wartości tych stosunków oraz czasu retencji poszczególnych substancji [1]. Ponieważ identyfikacja poszczególnych metabolitów często jest utrudniona, stosuje się dwie strategie. Jedna z nich polega na porównywaniu otrzymanego widma masowego z dostępnymi bibliotekami widm oraz bazami danych. Druga zaś polega na porównaniu widm związków wzorcowych z widmem uzyskanym podczas analizy [1].
Zarówno pomiary spektroskopowe, jak i spektrometryczne umożliwiają analizę metabolomu nie tylko pod względem jakościowym, ale także ilościowym. W obu tych kwestiach ważna jest tzw. standaryzacja pomiarów, a więc opracowanie odpowiednich protokołów dla pomiarów NMR, LC-MS, GC-MS czy innych. Standaryzacja, a więc jednakowe przygotowywanie prób (dla metody NMR), tzn. identyczne pH, stężenie buforu, wzorca (np. TSP) [13], objętość czy waga badanego materiału, temperatura jest niezwykle istotne w dalszej analizie statystycznej. Pozwala ona na eliminację błędów wynikających z różnej preparacji próbek, a tym samym skupić się na analizie i interpretacji różnic pomiędzy grupami, wynikającymi z istniejącego stanu rzeczy.
Analiza chemometryczna danych
Identyfikacja specyficznych związków chemicznych, biomarkerów, będąca jednym z głównych celów badań metabolicznych wymaga nie tylko niezwykłej staranności i powtarzalności w przygotowaniu próbek oraz wydajnych i czułych technik analitycznych, ale także zaawansowanych narzędzi chemometrycznych i statystycznych (rys. 2.) [14].
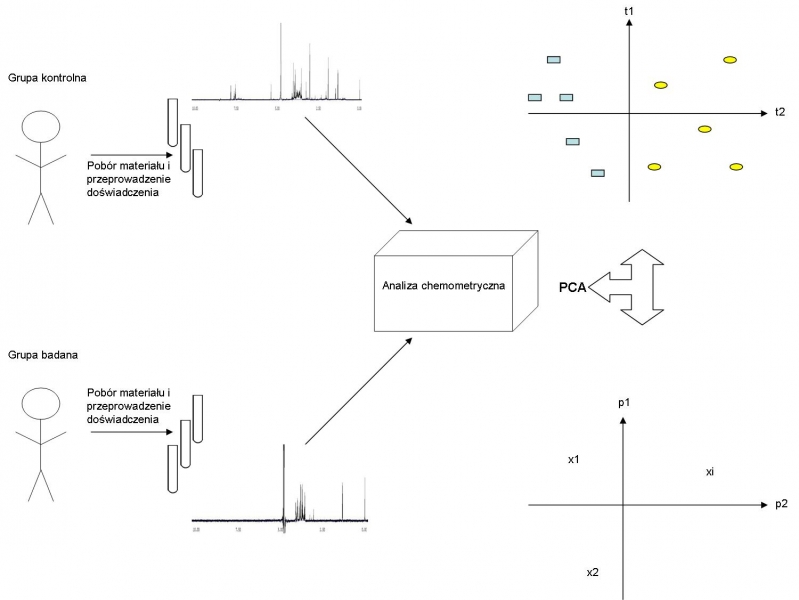
Rys. 2. Poszczególne etapy badań metabolomicznych
Podstawową metodą stosowaną w chemometrii, będącą punktem wyjścia do dalszych analiz jest analiza głównych składowych PCA (Principal Component Analysis) [15]. Jest to klasyczny przykład metody nienadzorowanej, służącej do projekcji, wizualizacji, wyznaczania trendu, a także identyfikacji wartości odstających we wszystkich obserwacjach lub próbach [16]. Ponieważ badania metabolomiczne obejmują badanie zbioru osób zdrowych (kontrola) oraz chorych, pozwala to na wyznaczenie relacji pomiędzy grupami, a także elementów, a więc potencjalnych biomarkerów, różnicujących obie grupy [15]. Dla opracowywania odpowiednich modeli, przewidywania niejednokrotnie subtelnych różnic oraz poszukiwania istotnych statystycznie biomarkerów często służą dwie inne metody, metoda częściowych najmniejszych kwadratów PLS (Partial least-squares) oraz ortogonalna metoda częściowych najmniejszych kwadratów OPLS (Orthogonal-PLS) [17-18]. Są to przykłady metod nadzorowanych, oparte na klasyfikacji poszczególnych obserwacji na podstawie dodatkowego parametru, jakim może być stan pacjenta (zdrowy, chory), rodzaj choroby, wiek czy płeć etc. [16-18].
Zastosowanie metod metabolomicznych w diagnostyce stanów chorobowych z przykładami
W obecnym czasie zastosowanie metod metabolomicznych w diagnostyce wszelkiego rodzaju chorób znajduje duże zainteresowanie wśród badaczy, co przekłada się na tysiące opublikowanych prac oryginalnych i przeglądowych. Poniżej zostaną przedstawione wybrane przykłady użycia metabolomiki w diagnostyce jedynie kilku stanów patologicznych.
Diagnostyka chorób nowotworowych
Rozwój i progresja wielu typów nowotworów musi znajdować swoje odzwierciedlenie w profilu metabolicznym płynów ustrojowych [19]. Bardzo istotnym problemem wśród dzieci oraz młodych ludzi jest kostniakomięsak, nowotwór złośliwy kości, dlatego umiejętne wykrywanie wczesnych zmian oraz zrozumienie zmian biochemicznych zachodzących podczas nowotworzenia może zredukować śmiertelność i przyczynić się do skrócenia terapii antynowotworowej [20].
Analizę profilu metabolitów moczu oraz surowicy krwi trzech grup badanych (pacjenci z łagodną zmianą nowotworową, złośliwą oraz kontrola) przeprowadzono wykorzystując metodę GC-MS. Zarówno mocz jak i surowicę krwi przed pomiarami poddano procesowi derywatyzacji [20].
W wyniku przeprowadzonych badań zidentyfikowano dwadzieścia dziewięć metabolitów w surowicy krwi oraz dwadzieścia jeden w moczu, a analiza chemometryczna pokazała wyraźnie rozdzielone trzy badane grupy. Wśród wszystkich metabolitów na uwagę zasługuje podwyższone stężenie cystyny (zarówno w moczu jak i w surowicy krwi) a obniżone fumaranu i malonianu (surowica krwi) oraz hipurynian (mocz) w obu grupach chorych w porównaniu z kontrolą. Podwyższone stężenie w surowicy u osób z kostniakomięsakiem zauważono dla kwasu 2-hydroksymasłowego (produkt uboczny konwersji metioniny do glutationu) oraz GABA zaś obniżone dla pirogronianu oraz mleczanu względem kontroli [20]. Badania te pokazują, że metabolomika z powodzeniem może być zastosowana jako nieinwazyjna metoda diagnostyczna oraz monitorująca przebieg choroby nowotworowej.
Główną przyczyną śmierci wśród kobiet na całym świecie jest nowotwór piersi. Pomimo prowadzonych programów antynowotworowych, zwiększających świadomość kobiet o konieczności przechodzenia badań przesiewowych liczba przypadków śmiertelnych jest wciąż bardzo duża [21]. Okresowe badania mammograficzne, badania palpilacyjne czy też testy krwi są niejednokrotnie niewystarczające, dają bowiem często fałszywie pozytywne wyniki i nie są w stanie wykryć dyskretnych zmian nowotworowych bądź też nawrotów, po wcześniejszej interwencji chirurgicznej. Poszukiwanie więc markerów metabolomicznych dla procesów nowotworzenia, stało się celem dla niektórych grup badawczych [21].
Asiago wraz ze współpracownikami przeprowadzili pomiary metabolomiczne surowicy krwi kobiet chorujących na raka piersi, wykorzystując metodę NMR oraz GCxGC-MS. Na uzyskanych w wyniku badań widmach oznaczono metabolity korzystając z dostępnych baz danych (dwadzieścia dwa metabolity NMR oraz osiemnaście GCxGC-MS) . Ostatecznie spośród wszystkich zidentyfikowanych związków wyselekcjonowano 11 metabolitów (siedem – NMR oraz cztery – GCxGC-MS) istotnych statystycznie [22]. Na podstawie tych danych stworzono model, który następnie został poddany kontroli. W tym celu grupę pacjentów po interwencji chirurgicznej monitorowano stosując konwencjonalną metodę immunologiczną oraz badania metabolomiczne wraz ze stworzonym modelem [22]. Okazało się, że znacząco szybciej (13 miesięcy wcześniej u około 55% pacjentów) proces nawrotu choroby wykryto korzystając z pomiaru profilu metabolicznego. Badania te pokazują, jaki potencjał diagnostyczny i predykcyjny może mieć pomiar profilu metabolicznego krwi osób chorych, a wykrycie nowotworu we wczesnej fazie rozwoju może skutecznie poprawić leczenie pacjentów [22].
Kolejnym przykładem wykorzystania narzędzi metabolomicznych w diagnozie chorób nowotworowych są badania przeprowadzone na pacjentach chorych na raka przewodu żółciowego [23, 24]. Okazuje się, że pomimo wysokiej specyficzności konwencjonalnych metod opartych na przeciwciałach nie są one stosunkowo czułe. Wyniki uzyskane z badań metabolomicznych wykazują znacznie wyższą nie tylko specyficzność, ale także bardzo wysoką czułość metody. Spośród metabolitów (cholina, kwas mlekowy, glukoza, kwas cytrynowy), na których skupiono główną uwagę w ocenie statystycznej, stwierdzono, że poziom kwasu cytrynowego u chorych jest znacznie wyższy niż w grupie kontrolnej. Logicznym wytłumaczeniem takiego stanu rzeczy wydaje się być fakt, że kwas cytrynowy poprzez acetylo-CoA jest prekursorem cholesterolu, który z kolei jest metabolizowany do kwasów żółciowych [24].
Powyższe przykłady pokazują, że nowatorska metoda jaką jest metabolomika bardzo skutecznie może przyczynić się do wczesnego diagnozowania nowotworów oraz monitorowania nawrotu choroby i postępów leczenia. W obecnym czasie badania metabolomiczne prowadzone na poziomie akademickim nie tylko pozwalają na rozróżnianie osób zdrowych od chorych na raka, ale również umożliwiają określenie ich fenotypów [25].
Profil metaboliczny chorób neurodegeneracyjnych i psychicznych
Badanie metabolomiczne wielu różnych neuroaktywnych związków dostarczyło wielu nieocenionych informacji o etiologii licznych chorób neurodegeneracyjnych i psychicznych, a tym samym przyczyniło się do poprawienia stanu wiedzy na temat diagnostyki tego typu schorzeń [26].
Bardzo trudną do diagnozy we wczesnym stadium, a jednocześnie bardzo powszechną formą demencji jest choroba Alzheimera (Alzheimer disease) [27]. W ostatnich latach zauważono również wzrost zainteresowania innym obrazem klinicznym, nazywanym łagodną utratą poznania MCI (mild cognitive impairment), uznawaną za wczesną formę choroby Alzheimera [28]. Niestety obecnie nie ma metody, która mogłaby rozróżnić oba schorzenia, a jednocześnie stwierdzić, czy pacjenci chorzy na MCI mogą w przyszłości zachorować na chorobę Alzheimera. Identyfikacja biomarkerów dla choroby Alzheimera oraz przedklinicznego syndromu MCI, a także monitorowanie progresji choroby jest bardzo istotna [28].
Greenberg wraz ze współpracownikami w swoich badaniach w poszukiwaniu biomarkerów różnicujących obie choroby wykorzystał metodę UPLC-MS, analizując profil metaboliczny surowicy krwi [29].
Analiza otrzymanych danych za pomocą PLS-DA pokazała wyraźne rozdzielenie wszystkich grup (kontrola – zdrowi, chorzy na AD i MCI). Na rozdział ten miał wpływ poziom trzech soli kwasów żółciowych, zidentyfikowanych jako GCD, GDCA oraz GCDCA. Niestety jak się okazało, podwyższony poziom tych metabolitów występuje zarówno w chorobie Alzheimera oraz MCI. Podobnie obserwacja wykresu PLS-DA pokazuje bliskie sąsiedztwo obu grup chorych w porównaniu z kontrolą [29].
Chociaż badania Greenberga nie pokazały bezpośredniego połączenia pomiędzy rozwojem chorób AD/MCI a poziomem metabolitów lipidowych, to jednak potwierdzają duża rolę związków lipidowych, homeostazy cholesterolu i metabolizmu b-amyloidu w chorobie AD [29].
Niezwykle śmiertelną chorobą neurodegeneracyjną, o skomplikowanej patogenezie, stawiającej wyzwanie dla wielu badaczy jest stwardnienie zanikowe boczne ALS (amyotrophic lateral sclerosis) [30]. Choroba ta należy do grupy chorób określanych jako chorobę neuronu ruchowego MND (motor neuron diseases) [30]. Metabolomiczną analizę surowicy krwi za pomocą metody NMR u chorych na ALS przeprowadził Kumar. W swoich badaniach oprócz grupy kontrolnej (zdrowej) użył kontroli dla chorych na ALS, którą stanowiły osoby chore na chorobę Hirayama (kontrolna choroba neurologiczna) [31].
W wyniku przeprowadzonych badań pokazano, że glutaminian, kwas b-hydroksymasłowy (BHBT), octan, aceton, kwas mrówkowy mają znacznie podwyższone stężenie u osób chorych na ALS, z kolei poziom glutaminy oraz histydyny był znacznie obniżony. Stężenia alaniny, lizyny pirogronianu, cytrynianu, glukozy, kreatyniny, kreatyninyny, oraz tyrozyny pozostawały bez zmian [31]. U osób chorych na chorobę Hirayama podwyższone stężenia istotnie statystyczne zanotowano dla glutaminianu, pirogronianu oraz kwasu mrówkowego, zaś BHBT, octan, aceton, alanina, lizyna, cytrynian, glukozy, kreatyniny, kreatyninyny, oraz tyrozyny pozostawały bez zmian [31]. Powyższe badania pokazują, że użycie metody NMR do wykrywania zmian zachodzących w profilu metabolicznym może być bardzo pomocne w późniejszym zastosowaniu diagnostycznym. Pomimo identyfikacji istotnie statystycznych metabolitów, wyjaśnienie etiologii stwardnienia zanikowego bocznego wymaga jeszcze wielu badań [31].
Wśród zastosowań badań metabolomicznych w medycynie należy wspomnieć o chorobach psychicznych. Bardzo często etiologia i patofizjologia tego typu schorzeń nie jest do końca znana, albo wielokrotnie jeszcze niepoznana [32]. Nadal główną metodą diagnostyczną chorób umysłowych jest wywiad kliniczny, rozmowa z biegłym psychiatrą. Poszukiwanie biomarkerów na różnego typu choroby jest więc istotne z punktu widzenia wczesnej diagnostyki oraz monitorowania postępów leczenia. Ponadto badania metabolizmu osób chorych umysłowo być może pozwolić na bliższe przyjrzenie się etiologii tych chorób [33].
Próbę znalezienia różnic w profilu metabolicznym osób zdrowych i chorych na schizofrenię podjął Holmes ze współpracownikami [34]. W swoich badaniach na płynie mózgowo rdzeniowym (PMR) trzech grup (chorzy leczeni, chory nieleczeni oraz kontrola) wykorzystał metodę NMR. Analiza danych spektroskopowych za pomocą PLS-DA pokazała wyraźną różnicę między trzema grupami, a identyfikacja metabolitów wskazała, że glukoza, octan, mleczan i glutamina są odpowiedzialne za rozdział grup.
Wyniki pokazują znaczne podwyższenie stężenia glukozy w płynie mózgowo rdzeniowym osób chorych nieleczonych w porównaniu z grupą kontrolną. Dodatkowo dla potwierdzenia tego faktu wykonano test na obecność glukozy w PMR u osób chorych nieleczonych, które potwierdziły wynik analizy metabolomicznej. Co ciekawe, badania na obecność glukozy we krwi nie pokazały żadnych różnic. Zakłócony metabolizm glukozy może wiązać się oczywiście ze zmianą nastroju oraz stanami psychotycznymi, jednakże podwyższone stężenie glukozy wraz z innymi zmianami w profilu metabolicznym (obniżone stężenie mleczanu i octanu) mogą stanowić specyficzne narzędzia w diagnostyce schizofrenii [34].
Analiza porównawcza osób chorych leczonych z kontrolą pokazuje, że około 50% pacjentów w wyniku leczenia przesuwa się w kierunku kontroli. To pozwala sugerować, że zastosowane leczenie jest skuteczne, a metabolomika jako metoda może być zastosowana do monitorowania tego procesu [34].
Diagnostyka nefrotoksyczności
Toksyczny wpływ leków na nerki jest spowodowany głownie przez funkcję jaką spełniają w organizmie. Praktycznie wszystkie toksyny oraz zbędne przemiany metabolizmu zawarte we krwi są usuwane wraz z moczem przez te właśnie organy. Monitorowanie więc oraz wczesna diagnoza nefrotoksyczności mogłaby przyczynić się do zmniejszenia przypadków uszkodzenia nerek w wyniku np. terapii lekowych [35].
Badanie wpływu różnych leków na nefrotoksyczność w swoich badaniach przeprowadził Boudonck wraz z współpracownikami. Do tego celu wykorzystał grupę zwierząt, szczurów, które leczone były cisplatyną, gentamycyną oraz tobramycyną oraz grupę kontrolną. Próby moczu oraz tkanek nerek zbierane były w ciągu kolejnych dni, a następnie analizowane przy pomocy LC-MS oraz GC-MS [36].
Analiza statystyczna otrzymanych danych (zidentyfikowanych metabolitów) pokazała znaczący wzrost stężeń aminokwasów w próbach moczu po 28 dniach od rozpoczęcia leczenia. W prawidłowo funkcjonującej nerce wszystkie aminokwasy ulegają resorpcji, natomiast nefrotoksyczność polekowa spowodowała znaczące podwyższenie poziomu aminokwasów (aminoaciduria). Oprócz wymienionych aminokwasów podwyższone stężenie wielu cukrów, tj. glukozy, mannozy, fukozy, kwasu N-acetylneuraminowego oraz glukonianu zauważono u trzech grup badanych zwierząt [36]. Poza cukrami oraz aminokwasami znaczący wpływ na różnicowanie grup badanych od kontroli miały wpływ inne metabolity, poliaminy, fosforan, 3-hydroksymaślan, 3-hydroksyfenylooctan, monoetanolamina i hipurynian [36]. Obserwacje dotyczące badań metabolicznych pobranych tkanek nerkowych pokazują natomiast obniżenie stężenia nukleozydów, będących prekursorami do syntezy nukleotydów [36].
Powyższe badania wykazały że metabolomika może być również użyteczną metodą w diagnozowaniu i monitorowaniu nefrotoksyczności. Być może w przyszłości pozwoli na szybszą i skuteczną diagnozę degeneracji nerek oraz innych narządów pod wpływem różnego rodzaju substancji, jak również przyczyni się do projektowania nowych, równie skutecznych, ale mniej toksycznych leków.
Podsumowanie
Metabolomika jest niezwykle użyteczną metodą, którą można z powodzeniem stosować w poszukiwaniu biomarkerów różnych stanów patologicznych, tj. różnego rodzajów nowotworów, choroby neurodegeneracyjne, stany psychiczne czy też w toksykologii. Umiejętne zrozumienie procesów metabolicznych jakie zachodzą w ludzkim organizmie jest bardzo ważne nie tylko dla zwiększenia skuteczności leczenia, ale także dla rozpoznawania choroby w jej zarodku, co z pewnością tę skuteczność znacznie poprawi. Dodatkowo metoda ta może pozwolić na stosowanie spersonifikowanego leczenia pacjentów.
Literatura
[1] Kraj A., Drabik A., Silberring J., Proteomika i metabolomika, Wydawnictwo Uniwersytetu Warszawskiego, 2010
[2] Greef J., Smilde A. K., Symbiosis of chemometrics and metabolomics: past, present, and future, J. Chemometrics, 2005, 19: 376–386
[3] Dembińska–Kieć A., Nastalski J.W., „Diagnostyka laboratoryjna z elementami biochemii klinicznej. Podręcznik dla studentów medycyny”, Elsevier Urban & Partner, Wrocław 2009.
[4] Chorąży M., Wprowadzenie do biologii systemów, NAUKA, 2011, 59-84
[5] Nordström A., Lewensohn R., Metabolomics: moving to the clinic, J. Neuroimmune Pharmacol, 2010, 5:4–17
[6] Wishart D. S., Tzur D., Knox C., Eisner R., Guo A. C., Young N., Cheng D., Jewell K., Arndt D., Sawhney S., Fung C., Nikolai L., Lewis M., Coutouly M. A., Forsythe I., Tang P., Shrivastava S., Jeroncic K., Stothard P., Amegbey G., Block D., Hau D. D., Wagner J., Miniaci J., Clements M., Gebremedhin M., Guo N., Zhang Y., Duggan G. E., Macinnis G. D., Weljie A. M., Dowlatabadi R., Bamforth F., Clive D., Greiner R., Li L., Marrie T., Sykes B. D., Vogel H. J., Querengesser L., HMDB: the Human Metabolome Database, Nucleic Acids Research, 2007, Vol. 35
[7] Beckonert O., Keun H. C., Ebbels T. M. D., Bundy J., Holmes E., Lindon J. C., Nicholson J. K., Metabolic profiling, metabolomic and metabonomic procedures for NMR spectroscopy of urine, plasma serum and tissue extracts, Nature Protocols, 2007, Vol. 2
[8] Bothwell J. H. F., Griffin J. L., An introduction to biological nuclear magnetic resonance spectroscopy, Biological Reviews, 2010
[9] Lei Z., Huhman D. V., Sumner W. L., Mass spectrometry strategies in metabolomics, The Journal of biological chemistry, 2011, Vol. 286
[10] Dettmer K., Aronov P. A., Hammock B. D., Mass spectrometry-based metabolomics, Mass Spectrometry Reviews, 2007
[11] Villas-Boˆas S. G., Mas S., A ˚kesson M., Smedsgaard J., Nielsen J., Mass spectrometry in metabolome analysis, Mass spectrometry reviews, 2005, Vol. 24
[12] Becker S., Kortz L., Helmschrodt C., Thiery J., Ceglarek U., LC–MS-based metabolomics in the clinical laboratory, Journal of Chromatography B, 883– 884, 2012, 68– 75
[13] Metz T.O., Metabolic Profiling, Springer Science+Business Media, LLC 2011
[14] Trygg J., Holmes E., Lundstedt T., Chemometrics in Metabonomics, Journal of proteome research, 2007, 6, 469-479
[15] Holmes E., Nicholls A. W., Lindon J. C., Connor S. C., Connelly J. C., Haselden J. N., Damment S. J., Spraul M., Neidig P., Nicholson J. K., Chemometric models for toxicity classification based on NMR spectra of biofluids, Chem. Res. Toxicol., 2000, 13, 471-478
[16] Gramacki J., Gramacki A., Wybrane metody redukcji wymiarowości danych oraz ich wizualizacji, XIV Konferencja PLOUG Szczyrk, 2008
[17] Bylesjo M., Rantalainen M., Cloarec O,, Nicholson J, K, Holmes E., Trygg J., OPLS discriminant analysis: combining the strengths of PLS-DA and SIMCA classification, Journal of Chemometrics, 2007, 20, 341-351
[18] Trygg J., Svante W., Orthogonal projections to latent structures (O-PLS), Journal of Chemometrics, 2002, 16, 119-128
[19] Kosliński P., Bujak R., Daghir E., Markuszewski M. J., Metabolic profiling of pteridines for determination of potential biomarkers in cancer disease, 2011, Electrophoresis, 32, 2044-2054
[20] Zhang Z., Qiu Y., Hua Y., Wang Y., Chen T., Zhao A., Chi Y., Pan L., Hu S., Li J., Yang C., Li G., Sun W., Cai Z., Jia W., Serum and urinary metabonomic study of human osteosarcoma, Journal of proteome research, 2010, 9, 4861-4868
[21] Nam H., Chung B. C., Kim Y., Lee KY., Lee D, Combining tissue transcriptomics and urine metabolomics for breast cancer biomarker identification, Bioinformatics, 2009, 25, 3151-3157
[22] Asiago V. M., Alvarado L. Z., Shanaiah N., Gowda G. A. N., Owusutu-Sarfo K., Ballas R. A., Raftery D., Early detection of recurrent breast cancer using metabolite profiling, Cancer research, 2010, 70, 8309-8318
[23] Issaq H. J., Fox S. D., Chan K. C., Veenstra T. D., Global proteomics and metabolomics in cancer biomarker discovery, Journal of Separation Science, 2011, 34, 3484–3492
[24] Wen H., Soo Yoo S., Kang J., Kim H. G., Park J-S., Jeon S., Lee J. I., Kwon H., N., Kang S., Lee D-H., Park S., A new NMR-based metabolomics approach for the diagnosis of biliary tract cancer, Journal of hepatology, 2010, 52, 228-233
[25] Yi Ng D. J., Pasikanti K. K., Yong Chan E. C., Trend analysis of metabonomics and systematic review of metabonomics-derived cancer marker metabolites, Metabolomics, 2011, 7, 155-178
[26] Caudle W. M. Bammer T. K., Lin Y., Pan S., Zhang J., Using ‘omics’ to define pathogenesis and biomarkers of Parkinson’s disease, Expert Rev Neurother, 2011, 10, 925-942
[27] Barba I, Fernandez-Montesinos R., Garcia-Dorado D., Pozo D., Alzheimer’s disease beyond the genomic era: nuclear magnetic resonance (NMR) spectroscopy-based metabolomics, Journal of cellular and molecular medicine, 2008, 12, 1477-1485
[28] Holmes E., Tsang T. M., Tabrizi S. J., The application of NMR-based metabonomics in neurological disorders, NeuroRx, 2006, 3, 358-372
[29] Greenberg N., Grassano A., Thambisetty M., Lovestone S., Legido-Quigley C., A proposed metabolic strategy for monitoring disease progression in Alzheimer’s disease, Electrophoresis, 2009, 30, 1235-1239
[30] Rozen S., Cudkowicz M. E., Bogdanov M., Matson W. R., Kristal B. S., Beecher C., Harrison S., Vouros P., Flarakos J., Vigneau-Callahan K., Matson T. D., Newhall K. M., Beal M. F., Brown R. H., Kaddurah-Daouk R., Metabolomic analysis and signatures in motor neuron disease, Metabolomics, 2005, Vol. 1
[31] Kumar A., Bala L., Kalita J., Misra U. K., Singh R. L., Khetrapal C. L., Babu G. N.,, Metabolomic analysis of serum by (1) H NMR spectroscopy in amyotrophic lateral sclerosis, Clinica Chimica Acta, 2010, Vol. 411
[32] Prabakaran S., Swatton J. E., Ryan M.M., Huffaker S. J., Huang Jt-J., Griffin J. L., Wayland M., Freeman T., Dudbridge F., Lilley K. S., Karp N. A., Hester S., Tkachev D., Mimmack M. L., Yolken R. H., Webster M. J., Torrey E. F., Bahn S., Mitochondrial dysfunction in schizophrenia: evidence for compromised brain metabolism and oxidative stress, Molecular Psychiatry, 2004, Vol. 9
[33] Orešič M., Tang J., Seppänen-Laakso T., Mattila I., Saarni S. E., Saarni S. I., Lönnqvist J., Sysi-Aho M., Hyötyläinen T., Perälä J., Suvisaari J., Metabolome in schizophrenia and other psychotic disorders: a general population-based study, Genome Medicine, 2011, Vol. 3
[34] Holmes E., Tsang Tsz. M., Huang J. T-J., Leweke F. M., Koethe D., Gerth C. W., Nolden B. M., Gross S., Schreiber D., Nicholson J. K., Bahn S., Metabolic profiling of CSF: evidence that early intervention may impact on disease progression and outcome in schizophrenia, PLoS medicine, 2006, Vol. 3
[35] WeissR. H., Kim K., Metabolomics in the study of kidney disease, Nature reviews. Nephrology, 2012, 8, 22-33
[36] Boudonck K. J., Mitchell M. W., Nemet L., Keresztes L., Nyska A., Shinar D., Rosenstock M., Discovery of metabolomics biomarkers for early detection of nephrotoxicity, Toxicologic Pathology, 2009, 37, 280-292
* Wydział Chemii, Politechnika Wrocławska, Wybrzeże Wyspiańskiego 27, 50-370 Wrocław

